"Die große Stärke beim Repurposing ist der Faktor Zeit"
| 05. Mai 2020Auf ein neues Medikament gegen COVID-19 muss man wohl noch Jahre warten. Währenddessen untersuchen Wissenschafter*innen bereits bekannte Arzneistoffe auf ihre Wirksamkeit gegen das Virus. Gerhard Ecker, Dekan der Fakultät für Lebenswissenschaften, erklärt, wo die Stärken des "Repurposing" liegen.
uni:view: Herr Ecker, warum ist es so schwierig, COVID-19 mit Arzneistoffen zu behandeln?
Gerhard Ecker: Viren sind sehr speziell, da sie für ihre Vermehrung eine Wirtszelle befallen müssen. Die Zelle wird dann so umprogrammiert, dass sie all ihre Energie in die Herstellung neuer Viruspartikel investiert. Mit dem Verständnis dieser Prozesse ergeben sich die möglichen Angriffspunkte für Arzneistoffe: Das Andocken des Virus an seine Wirtszelle erfolgt zum Beispiel über ein sogenanntes "Spike-Protein" auf der Oberfläche des Virus, das mit einem ACE-Protein auf der Oberfläche der Lungenzellen interagiert.
Nach Eindringen in die Zelle werden Proteine wie zum Beispiel die RNA-abhängige RNA-Polymerase freigesetzt, die die Wirtszelle umprogrammieren. Diese Proteine sind der Angriffspunkt vieler bekannter antiviraler Arzneistoffe. Das Grippemittel Tamiflu etwa verhindert das Ausschleusen der Viruspartikel aus der Zelle. Allerdings ist Tamiflu nur in den ersten Tagen der Infektion wirksam, da merken die meisten noch gar nichts von COVID-19.
uni:view: Immer wieder heißt es, dass die Entwicklung eines neuen COVID-19-Medikaments viele Jahre dauern wird. Können Sie das erklären?
Ecker: Die Entwicklung eines neuen Arzneistoffes ist ein sehr komplexer Prozess. Es geht nicht nur um Wirkung, Wirkdauer und Stabilität im Körper, es müssen auch mögliche Nebenwirkungen erkannt und minimiert werden. Dies erfordert je nach Ausgangslage die Testung von Millionen bekannter chemischer Verbindungen sowie die Synthese tausender neuer Substanzen. Die Erfolgschancen liegen deutlich unter fünf Prozent, die Kosten liegen bei mehreren Milliarden Euro, und das ganze dauert zehn bis 15 Jahre. Für die aktuelle COVID-19-Pandemie ist eine Neuentwicklung also keine Option. Eine Möglichkeit, das langwierige Verfahren deutlich abzukürzen, ist die Verwendung bereits vorhandener Arzneistoffe, das sogenannte Repurposing oder Repositioning.
uni:view: Wo liegen die Vorteile des Repurposing?
Ecker: Die große Stärke beim Repurposing ist der Faktor Zeit. Bei einem Arzneistoff, der sich bereits am Markt befindet, kennt man die Verträglichkeit und Nebenwirkungen. Die umfangreichen preklinischen Studien sowie die Phase 1 der klinischen Studien fallen weg, man kann sofort die Wirksamkeit testen. Grundsätzlich gibt es zwei Ansätze: die Virusinfektion zu bekämpfen oder die Symptome zu bekämpfen.
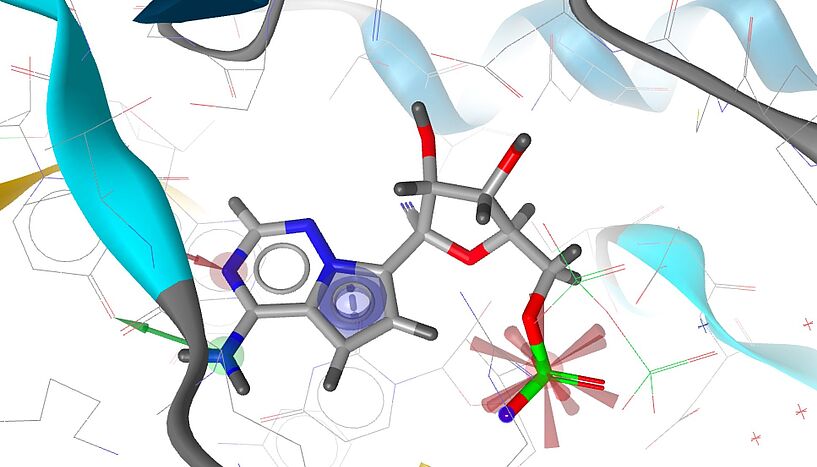
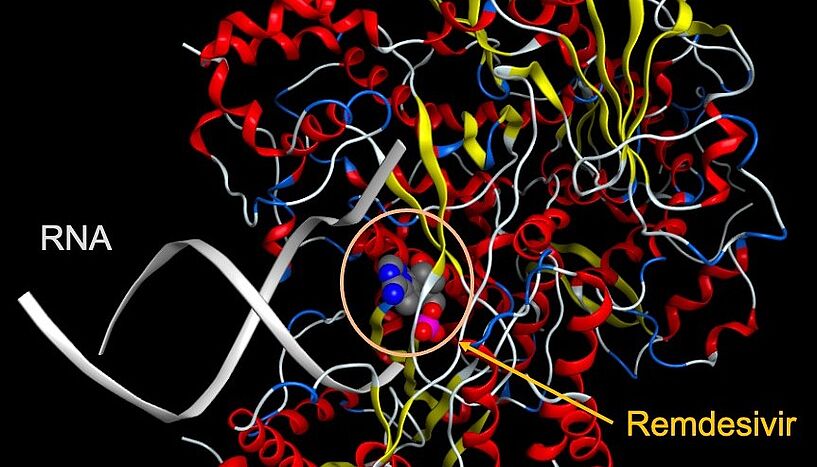
Als Repurposing-Kandidaten gegen SARS-CoV-2 eignen sich in erster Linie antivirale Arzneistoffe, wie zum Beispiel Ritonavir, Lopinavir oder Remdesivir. Zur Bekämpfung der Symptome von COVID-19 wurden auch die Antimalariamittel Hydroxychloroquin und Chloroquin getestet, haben sich aber nicht bewährt. Insgesamt laufen aktuell mehr als 200 klinische Studien mit verschiedensten Verbindungen. Ein anderer Weg wäre die Verwendung von Verbindungen, die zwar noch nicht am Markt sind, aber zumindest Phase 1 bestanden haben. Hier wird immer wieder Remdesivir genannt, das die Virusvermehrung in der infizierten Zelle stoppt.
Corona-Virus: Wie es unser Leben verändert
Von neuen familiären Abläufen bis hin zu den Auswirkungen auf Logistikketten: Expert*innen der Universität Wien sprechen über die Konsequenzen des Corona-Virus in unterschiedlichsten Bereichen. (© iXismus/Pixabay)
Zum Corona-Dossier
uni:view: Wie wirkt Remdesivir?
Ecker: Remdesivir wurde ursprünglich gegen Ebola-Infektionen entwickelt und wurde auch bei anderen RNA-Viren wie SARS und MERS getestet. In den USA wurde Remdesivir am 1. Mai in einem Eilverfahren zur Behandlung schwerer Coronafälle zugelassen. Es hemmt die RNA-abhängige RNA-Polymerase und reduziert damit die Produktion neuer Viren.
Ein anderer interessanter Ansatz ist die Verbindung APN01 der Firma Apeiron, die das Andocken des Virus an die Lungenzellen blockiert. Das Corona-Virus dockt über ein Spike-Protein an ACE2-Rezeptoren an der Wirtszelle an. APN01 sieht aus wie ACE2 und heftet sich an das Spike-Protein des Virus. Vielleicht hilft dieses Bild beim Verständnis: Stellen Sie sich eine Anhängerkupplung bei einem Auto vor – das ist das Virus – und einen aufgeschnittenen Tennisball, der als Schutz auf der Kupplung sitzt und im Grunde einen Anhänger imitiert. Die Arzneistoffe nehmen die Rolle des Tennisballs ein und Verhindern das "Ankuppeln" an die Zelle.
uni:view: Wo kommt das Repurposing von Arzneistoffen an seine Grenzen?
Ecker: Man hat beim Repurposing leider keinerlei Spielraum, wenn es um die Anpassung an die Krankheit geht. Entweder man hat das Glück, dass eine der Verbindungen gut genug wirkt, oder man muss ganz von vorne beginnen. Dies ist einer der Gründe, warum sehr große Anstrengungen in die Entwicklung eines Impfstoffes gehen – doch selbst dieser Prozess kann Jahre dauern. Man muss in Bezug auf Repurposing auch realistisch sein: Wenn einer der gängigen antiviralen Arzneistoffe Corona-Infektionen zuverlässig verhindern könnte, würden wir es vermutlich schon wissen.
uni:view: Wie beurteilen Sie den internationalen Forschungsprozess in dieser Ausnahmesituation?
Ecker: Sehr positiv ist, dass das Genom bereits im Jänner nach der Isolierung des Virus veröffentlicht wurde, inzwischen gibt es mehr als 100 3D-Strukturen von Virusproteinen in der Public Domain. Dabei handelt es sich um eine noch nie dagewesene Transparenz von Daten, die die Forschung natürlich sehr beschleunigt. So kennt man zum Beispiel die Strukturen der Proteine, die in den ersten Schritt einer Infektion involviert sind, schon sehr genau. Auch die Proteine, die für das Umprogrammieren verantwortlich sind, wurden mittlerweile identifiziert.
Vielen Dank für das virtuelle Gespräch! (bw)
Gerhard Ecker ist Dekan der Fakultät für Lebenswissenschaften und leitet die Pharmacoinformatics Research Group am Department für Pharmazeutische Chemie. Zu seinen Forschungsschwerpunkten zählen die Entwicklung von Computermodellen zur Vorhersage von Nebenwirkungen, die Analyse der molekularen Basis der Interaktion von Arzneistoffen mit Transportproteinen, sowie das Analysieren und Verknüpfen großer Datenbanken im Bereich Lebenswissenschaften. Er ist zudem wissenschaftlicher Leiter des Zertifikatskurses „Klinische Pharmazie – Medikationsanalyse“. ( © Barbara Mair)