Salmonellen: Was nach der Infektion in der Zelle passiert
| 05. Februar 2016
ForscherInnen der Universität Wien und der Universität Würzburg haben die Genexpression von Salmonellen-infizierten Zellen sowie jene der Salmonellen gleichzeitig untersucht und dabei Überraschendes festgestellt. (Foto: Rocky Mountain Laboratories, NIAID, NIH)
Wenn Salmonellen menschliche Zellen infizieren, werden viele kleine Regler in den Bakterien aktiv. Das fanden Wissenschafter der Universität Wien gemeinsam mit Würzburger KollegInnen heraus und publizierten dazu in "Nature". Was dieses Ergebnis konkret bedeutet, erklären sie in uni:view.
Salmonellen sind Bakterien, die überall in der Umwelt vorkommen – prinzipiell kann jedes Nahrungsmittel Salmonellen enthalten. Beim Menschen können die stäbchenförmigen Bakterien schwere Magen-Darm-Erkrankungen hervorrufen. Was nach einer Infektion genau passiert, haben Fabian Amman vom Department für Chromosomenbiologie und Peter Stadler vom Institut für Theoretische Chemie der Universität Wien gemeinsam mit KollegInnen aus Würzburg untersucht. Dafür haben sie menschliche Zellen in Kulturschalen von Salmonellen befallen lassen und die regulatorischen RNAs von beiden gleichzeitig sequenziert.
Duale RNA-Sequenzierung
Das war aufgrund der sogenannten "Dualen RNA-Sequenzierung" möglich – eine Methode, die bis vor kurzem noch schwer realisierbar war. "Es ist uns gelungen, die Genexpression Bakterien-infizierter Wirtszellen – gleichzeitig mit der des bakteriellen Erregers, in unserem Fall Salmonellen –, zu messen", erklärt es Alexander Westermann von der Universität Würzburg. Mitautor Fabian Amann von der Universität Wien ergänzt: "Durch den technologischen Fortschritt konnten wir nun seltene RNA-Moleküle aus Bakterien und sehr häufige RNA-Moleküle in den humanen Wirtszellen gleichzeitig bestimmen, quantifizieren und in Relation zueinander zu setzen."
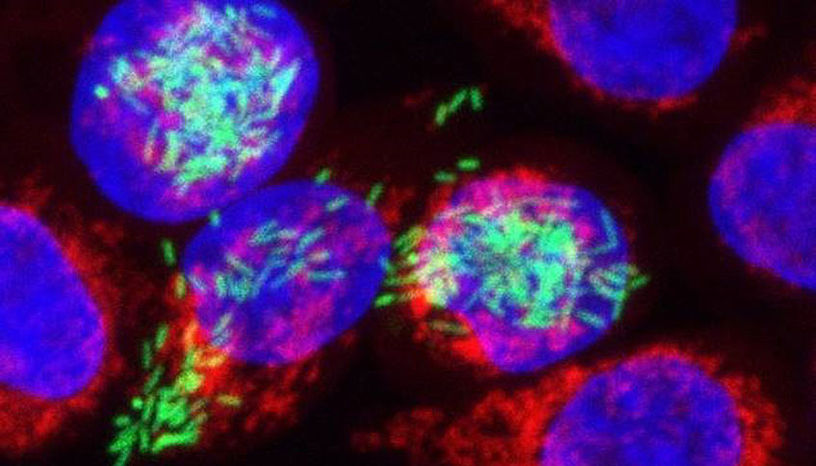
Das Bild zeigt eine Fluoreszenzmikroskopieaufnahme von Salmonellen-infizierte HeLa-Zellen. Die hier verwendeten Salmonellen entleeren ein fluoreszierendes Protein und erscheinen deshalb grün. Die humanen Wirtszellen wurden mit fluoreszierenden Farbstoffen gefärbt (rot/blau).
Fleißige Regler
Spannend ist auch, was nach der Infektion passiert: Die Bakterien wurden nämlich erst mit der Zeit so richtig aktiv. Während nach vier Stunden gerade einmal 1,4 Prozent der regulatorischen RNAs von ihnen stammten, waren es nach einem Tag 13,7 Prozent, berichten die ForscherInnen in "Nature". Von den über 300 regulatorischen RNAs der Salmonellen waren manche um das Zehnfache präsenter, eine sogar um das Hundertfache. Diese besonders fleißigen Regler nennen sich "PinT". Sie sorgen für das nötige Timing, wenn sich die Salmonellen nach der Invasion in den Wirtszellen breit machen, und schwächen deren Abwehrreaktionen.
Überrascht war das deutsch-österreichische ForscherInnenteam davon, dass im Laufe einer Salmonelleninfektion eine einzige RNA die Expressionsrate von ungefähr zehn Prozent aller RNA-Moleküle des Wirts beeinflussen kann.
Wie sich Bakterien etablieren
"PinT" formt vermutlich den Übergang vom invasiven Status der Mikroben zu einem Zellprogramm, mit dem sie sich optimal in den Wirtszellen vermehren können. Der "fleißige Regler" manipuliert aber auch die betroffenen menschlichen Zellen, fanden die WissenschafterInnen heraus. So unterdrückt er etwa Botenstoffe, die eine Entzündungsreaktion und damit die zelluläre Abwehr unterstützen. Dadurch können sich die Bakterien besser in den Zellen etablieren, meinen die ForscherInnen. Mit ihrer Arbeit zeigen sie, welche Auswirkungen die Regulation weniger, spezifischer Target-mRNAs im Bakterium auf die Wirtsantwort haben kann.
Ansätze für neue Therapien
Die neue Methode bietet die Möglichkeit, Infektionen aus einem neuen Blickwinkel zu studieren. In Zukunft könnten neue molekulare Schnittstellen der Wirt-Pathogen-Interaktion aufgezeigt werden, von denen einige vielleicht sogar neue Ansatzpunkte für Therapiestrategien bieten.
Mit diesem universalen Ansatz ist es außerdem möglich, das Verhalten von Krankheitserregern umfangreich und genau zu testen. "Unsere Methode kommt der wirklichen Umgebung während einer Krankheit näher als viele bisherige Tests. Sie ermöglicht es im Detail zu verstehen, wie sich Wirt und Pathogen gegenseitig beeinflussen", erklärt Fabian Amman.
Tests werden einfacher und billiger
Ihm zufolge können solche Ansätze langfristig dabei helfen, den Phenotyp von Pathogenen zu bestimmen. "Bisher führte man die Eigenschaften neu auftretender Krankheiterregerstämme meist auf den Genotyp zurück. Dabei wurde die Regulation entscheidender Gene oftmals außen vorgelassen. Mit der 'Dualen RNA-Sequenzierung' hingegen kann man relativ einfach und billig testen, in welchen Eigenschaften sich neue Stämme von bereits bekannten unterscheiden", schließt Fabian Amman. (APA/ps)
Das Paper "Dual RNA-seq unveils noncoding RNA functions in host–pathogen interactions" (AutorInnen: Alexander J. Westermann, Konrad U. Förstner, Fabian Amman, Lars Barquist, Yanjie Chao, Leon N. Schulte, Lydia Müller, Richard Reinhardt, Peter F. Stadler und Jörg Vogel) erschien am 20. Jänner 2016 in "Nature".