Mikrobenjagd mit schwerem Wasser
| 30. Dezember 2014Mithilfe von schwerem Wasser (D2O) haben MikrobiologInnen der Universität Wien gemeinsam mit internationalen KollegInnen erstmals die Stoffwechselaktivitäten einzelner Mikrobenzellen verfolgt und gezielt genomisch charakterisiert.
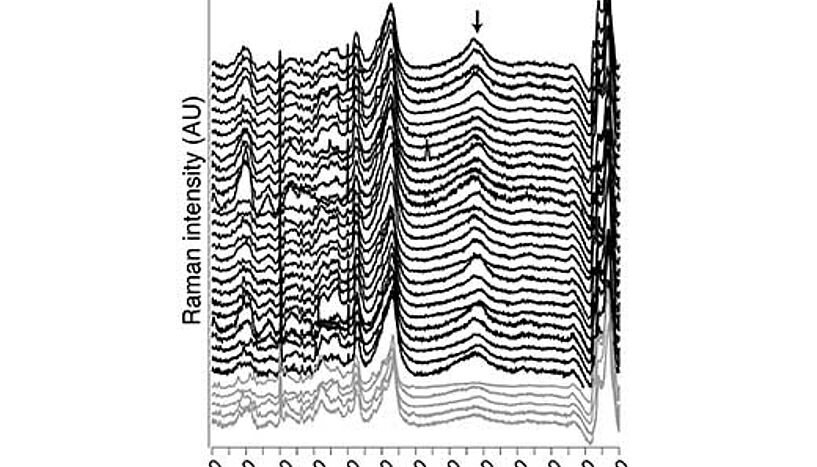
Die WissenschafterInnen versorgten die Mikroorganismen mit Wasser, das statt herkömmlichem Wasserstoff (H) das etwa doppelt so schwere Wasserstoff-Isotop Deuterium (D) eingebaut hatte. Damit konnten sie die Stoffwechselaktivitäten einzelner Mikrobenzellen aus dem Mäusedarm in Anwesenheit verschiedener Energiequellen verfolgen. "Stoffwechselaktive Mikroben verwenden dieses Deuterium für den Aufbau ihrer Fett- und Eiweißstoffe und werden so durch einen chemischen Fingerabdruck mittels Raman-Mikrospektrometrie oder NanoSIMS identifizierbar", erklärt Michael Wagner, Leiter des Departments für Mikrobielle Ökologie und Leiter der Studie.
Aktive Zellen aussortiert und genomisch analysiert
Besonders spannend an dieser Methode ist, dass die mit Deuterium markierten Zellen nach der Untersuchung im Raman-Mikrospektrometer einzeln – mithilfe einer optischen Pinzette – aussortiert und dann genomisch untersucht werden können. "Damit ist es erstmals möglich, gezielt Genome einzelner Mikrobenzellen mit einer bestimmten Funktion in einem Ökosystem zu untersuchen", ergänzt David Berry, Assistenzprofessor am Department und Erstautor der Studie. Besonders hilfreich ist auch, dass die Zugabe von schwerem Wasser die chemische Zusammensetzung der Probe kaum beeinflusst.
Neue Mikroben entdeckt und Aktivitätsmuster charakterisiert
Mit dieser neu entwickelten Methode entdeckten die ForscherInnen verschiedene, bisher noch nicht identifizierte Mikroben, die im Mäusedarm Glucosamin bzw. Mucin abbauen. Darüber hinaus charakterisierten sie die Aktivitätsmuster von zwei der im Mäusedarm vorherrschenden Bakterienarten.
Weiterentwicklung der Methode für Medizin und Umwelt gefragt
Aufbauend auf diesen Ergebnissen arbeiten Mikrobiologe Wagner und sein Team nun daran, den Durchsatz dieser Technik zu erhöhen. "Mithilfe eines Mikrofluidic-Chips, der zusammen mit einem Raman-Mikrospektrometer vor ein FACS-Gerät geschaltet werden soll, hoffen wir, schon bald halbautomatisch tausende einzelner Mikrobenzellen mit einer gewünschten Funktion aus medizinischen oder Umweltproben für genomische Analysen aussortieren zu können", so Wagner. (APA/red)
Das Paper "Tracking heavy water (D2O) incorporation for identifying and sorting active microbial cells" (AutorInnen: David Berry, Esther Mader, Tae Kwon Lee, Dagmar Woebken, Yun Wang, Di Zhu, Marton Palatinszky, Arno Schintlmeister, Markus C. Schmid, Buck T. Hanson, Naama Shterzer, Itzhak Mizrahi, Isabella Rauch, Thomas Decker, Thomas Bocklitz, Jürgen Popp, Christopher M. Gibson, Patrick W. Fowler, Wei E. Huang und Michael Wagner) erschien am 29. Dezember 2014 im Journal "Proceedings of the National Academy of Sciences (PNAS).