"Meine Forschung": RNA-Microarrays
| 22. März 2019
Chemikerin Kathrin Hölz kopiert im Rahmen ihrer Dissertation molekularbiologische Vorgänge des Körpers, um Gene gezielt zu regulieren. (© sciencephoto/Shutterstock.com)
Können Gene gezielt reguliert werden? Das erforscht Chemikerin Kathrin Hölz von der Universität Wien. Sie entwickelt Methoden zur enzymatischen Herstellung von RNA-Microarrays, die bei der Untersuchung von "genetischen Schaltern" helfen. Diese könnten u.a. den Ausbruch von Krankheiten verhindern.
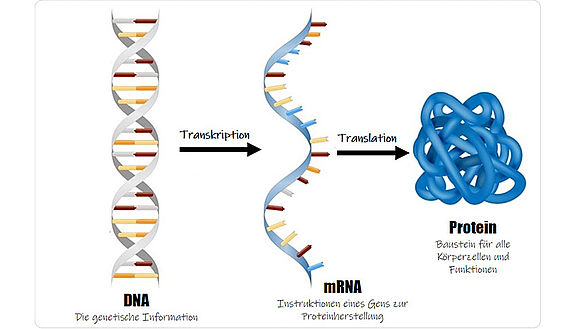
Gene sind Abschnitte der DNA, die Grundinformationen sowohl für die Entwicklung von individuellen Eigenschaften als auch zur Herstellung biologisch relevanter Moleküle enthalten. Ist ein Gen aktiv, wird durch die sogenannte Transkription dieses Gens die Boten-RNA (messenger RNA, mRNA) hergestellt, welche wiederum weitere biologische Vorgänge in Gang setzt.
Ist ein Gen passiv, entsteht keine mRNA, wodurch auch keine biologischen Prozesse eingeleitet werden, die in weiterer Folge zur Herstellung von Proteinen und zur Ausbildung von Eigenschaften führen könnten. Einblicke in die Genexpression liefern einen allgemeinen Überblick darüber, welche Gene aktiv und welche passiv sind. Aber kann diese Expression auch beeinflusst werden? Können Gene gezielt angesteuert und reguliert werden?
Vom Gen zum Protein: Die DNA ist der Träger der genetischen Information. Gene sind bestimmte Abschnitte der DNA. Während der Transkription werden solche Abschnitte abgelesen und einzelsträngige Kopien, die Boten-RNAs (messenger RNA, mRNA), hergestellt. Diese mRNAs enthalten Instruktionen zur Herstellung von Proteinen. Im Prozess der Translation werden die Instruktionen der mRNAs ausgelesen und die Proteine entsprechend hergestellt. (© adapted from Designua/Shutterstock.com)
"Genetische Schalter"
Tatsächlich widmen sich viele molekularbiologische Forschungsgruppen der Entwicklung sogenannter synthetischer Genschalter, welche es erlauben, Gene gezielt zu regulieren, um somit zum Beispiel den Ausbruch von Krankheiten zu verhindern, bestimmte Abläufe im Körper zu steuern oder spezifische Gentherapeutika zu entwickeln.
Darüber hinaus wurde festgestellt, dass mRNA-Moleküle in Bakterien sogar selbst Regionen zur Regulierung der Genexpression enthalten – so genannte Riboswitches. Binden diese natürlich vorkommenden "Schalter" an bestimmte Zielverbindungen, ändert sich ihre Struktur. Diese Strukturänderung hat wiederum direkten Einfluss auf die Aktivität des Gens, welches die entsprechende Boten-RNA kodiert. Doch wie lassen sich diese Wechselwirkungen genauer untersuchen und in weiterer Folge nutzbar machen?
Im uni:view-Dossier "Meine Forschung" stellen DoktorandInnen der Universität Wien ihre Forschungsprojekte vor. Das Dossier läuft in Kooperation mit dem DoktorandInnenzentrum. (© Universität Wien)
Microarrays und ihre Herstellung
Microarrays sind wichtige biotechnologische Werkzeuge, die sich für solche Untersuchungen hervorragend eignen. Standardmäßig werden sie vor allem in der molekularbiologischen Analytik eingesetzt, da sie ForscherInnen ganze Genomanalysen in einzelnen Experimenten ermöglichen. Microarrays bestehen aus einem festen Träger, dem Chip, der beispielsweise aus Glas bestehen kann, und einer Vielzahl darauf gebundener DNA- oder RNA-Stränge.
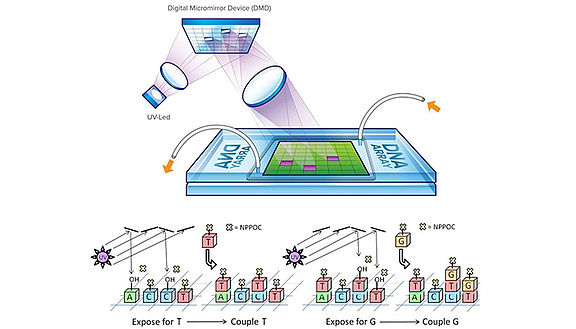
Sie lassen sich auf verschiedene Weise herstellen (Spotting, Microprinting, Multielektroden, Photolithographie), wobei grundsätzlich unterschieden wird, ob komplette kurzsträngige DNA-Abschnitte auf der Chipoberfläche befestigt werden oder der Microarray durch in situ Synthese, also den schrittweisen Aufbau von Nukleinsäuresträngen, ähnlich dem Prinzip von Lego-Türmen, hergestellt wird. Hierbei bietet vor allem die lichtgesteuerte photolithographische in situ Synthese den großen Vorteil, dass sie zum einen sehr flexibel bezüglich des Microarraydesigns ist und zum anderen eine viel höhere Dichte an Sequenzen auf der Chipoberfläche zulässt, was vor allem in der späteren Anwendung von enormem Nutzen ist.
Bei der photolithographischen in situ Synthese von DNA Microarrays wird eine Mikrospiegelvorrichtung (Digital Micromirror Device, DMD) genutzt, um UV-Licht in einem bestimmten Muster auf die Chipoberfläche zu projizieren. Dort befinden sich die Synthesebausteine, deren photolabile Schutzgruppe durch Lichteinfall abgespalten wird. Da jeder Mikrospiegel gezielt angesteuert werden kann, ist hierdurch die gleichzeitige Synthese von hunderttausenden unterschiedlichen DNA-Strängen möglich. (© Mark M. Somoza)
Gezielter Einsatz von Enzymen
Obwohl die Herstellung von DNA-Microarrays also längst etabliert ist, lässt sich diese Methode trotzdem nur bedingt auf die Synthese der gewünschten RNA-Microarrays übertragen. RNA-Moleküle sind deutlich instabiler und erfordern daher eine besonders vorsichtige und sorgfältige Arbeitsweise.
Das Hauptproblem in der Synthese von RNA-Microarrays liegt allerdings darin, dass die schrittweise Bindung von einzelnen RNA-Bausteinen an den wachsenden RNA-Strang eine geringere Effizienz aufweist als ihre DNA-Äquivalente. Hierdurch wird die Länge der RNA-Sequenzen auf der Microarrayoberfläche limitiert. Die angestrebten, oben erwähnten Anwendungen, erfordern allerdings die Herstellung deutlich längerer RNA-Stränge als zum jetzigen Zeitpunkt mit der chemischen Synthese von RNA-Microarrays möglich ist.
Von DNA-Microarrays zu RNA-Microarrays
In meiner Dissertation verfolge ich daher den Ansatz, molekularbiologische Vorgänge des Körpers zu kopieren, um DNA-Microarrays durch in vitro Transkription in RNA-Microarrays umzuwandeln. Hierbei nutze ich im Anschluss an die lichtgesteuerte in situ Synthese von DNA-Microarrays spezielle Enzyme, um die DNA-Vorlagen längenunabhängig in ihre komplementären RNA-Stränge umzuschreiben.
Die Anwendung weiterer Enzyme ermöglicht den selektiven Abbau der DNA-Vorlagen, so dass anschließend ein reiner RNA-Microarray erhalten wird. Mit dieser Methodenentwicklung zur Herstellung von RNA-Microarrays wird eine wichtige Grundlage für spätere Anwendungsmöglichkeiten geschaffen.
Kathrin Hölz, geb. 1987 in Bingen am Rhein, hat Chemie an der Johannes Gutenberg-Universität Mainz studiert. Ihre Dissertation mit dem Thema "Approaches to the enzymatic synthesis of high-density RNA microarrays" führt sie an der Fakultät für Chemie der Universität Wien unter der Betreuung von Mark M. Somoza durch. (© PicturePeople/Kathrin Hölz)
Literaturtipps zum Thema:
- "Advances in Light-Directed Synthesis of High-Density Microarrays and Extension to RNA and 2ʹF-ANA Chemistries" von J. Lietard, M. J. Damha und M. M. Somoza in Enzymatic and Chemical Synthesis of Nucleic Acid Derivatives, herausgegeben von Jesús Fernandéz Lucas und María-José Camarasa Rius, Wiley-VCH Verlag.
- "Der Experimentator: Microarrays" von H.-J. Müller und T. Röder, Spektrum Akademischer Verlag.