Der Protein-Netzwerker
| 19. April 2013
Thomas Rattei, Leiter des Departments für Computational Systems Biology an der Fakultät für Lebenswissenschaften, bei der Arbeit.
Was haben die Erreger menschlicher Erkrankungen, mikrobielle Ökosysteme, Ameisen, Amöben und "Ötzi" gemeinsam? Sie alle waren schon Thema in Projekten von Thomas Rattei, Leiter des Departments für Computational Systems Biology. Ein Interview über die Querschnittsdisziplin Bioinformatik.
Ohne Proteine geht auf dieser Welt rein gar nichts: Sie bauen die Zellen aller Organismen auf, sie sind es, die Signale mit anderen Zellen und der Umwelt austauschen. Allein können Proteine aber nichts ausrichten. Erst ihre Vernetzung macht Lebewesen lebensfähig. Der Bioinformatiker Thomas Rattei von der Fakultät für Lebenswissenschaft erforscht die Evolution dieser Proteinnetzwerke. Gemeinsam mit dem Physiker Hernan Makse von der City University New York (CUNY) hat er jetzt in der renommierten Fachzeitschrift PLOS ONE seine Erkenntnisse veröffentlicht. Im Interview erklärt er, was er tut.
Sie sagen "Das Wissen um Funktion und Evolution von Proteinnetzwerken ist aktuell eine der spannendsten Fragen in der Biologie". Warum?
Thomas Rattei: Die molekulare Datenflut der letzten beiden Jahrzehnte hat die Biologie vor die für sie neue Aufgabe gestellt, biomolekulare Informationen in großer Menge zu interpretieren, daraus Theorien abzuleiten und mit diesen wiederum Vorhersagen zu generieren. Wir wissen, dass die meisten Proteine in unseren Zellen miteinander wechselwirken müssen, um ihre Aufgaben zu erfüllen. Wie genau sie das tun, ist noch sehr wenig bekannt. Dennoch liegt darin ein wichtiger Schlüssel z.B. zur Vorhersage des Krankheitsrisikos einer Mutation in einem menschlichen Gen.
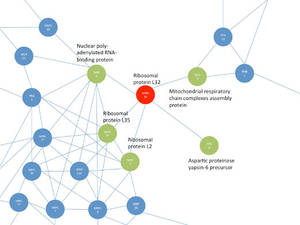 | Ausschnitt eines Proteinnetzwerkes (zum Vergrößern anklicken) |
|---|
Obwohl die Biologie in diesem Punkt deutlich komplexer ist als z.B. die Elementarteilchenphysik, hilft uns hier der Prozess der Evolution. Dieser erlaubt es uns, aus dem Vergleich verschiedener Organismen Rückschlüsse auf die Funktion von Biomolekülen zu ziehen. Dies gilt ganz besonders für die Proteinnetzwerke, deren direkte Messung sehr zeit- und kostenintensiv ist. Mit Hilfe der "Vergleichenden Genomik" können wir unter Berücksichtigung der evolutionären Prozesse zahlreiche sehr wertvolle Vorhersagen über die Funktion von Proteingruppen machen, die noch nie direkt gemessen wurden.
In Ihrer aktuellen Publikation in PLOS ONE vergleichen Sie so genannte "rekonstruierte Proteinnetzwerke". Was darf man sich darunter vorstellen?
Rattei: Die Evolution der Proteinnetzwerke vollzieht sich über lange Zeiträume und ist mangels molekularer Fossilien nicht direkt beobachtbar. Daher haben wir aus heute beobachtbaren Daten, die hauptsächlich aus Genomanalyse-Projekten stammen, die wahrscheinlichsten Proteinnetzwerke der frühen Vorfahren unserer heutigen Arten vorhergesagt. Einige der dafür notwendigen Computer-Methoden sind in der Bioinformatik bereits sehr bewährt. Andere Teile der Methodik wurden von unseren KollegenInnen in New York und von uns neu entwickelt.
 | Am beschriebenen Paper beteiligt (v.l.n.r.): Thomas Weinmaier (Dissertant), Thomas Rattei (Projektleiter), Dmitrij Turaev (Dissertant). |
|---|
Sie suchen also den Bauplan von Proteinnetzwerken. Wie geht man so etwas an? Bitte erzählen Sie etwas über Ihre tägliche Arbeit – findet die vornehmlich im Labor statt, am Computer, im Kopf?
Rattei: Unsere Arbeitsgruppe arbeitet seit etwa zehn Jahren ausschließlich am Computer. Wir analysieren biologische Daten, entwickeln neue Theorien, programmieren Datenbanken und Vorhersageprogramme oder verfeinern bioinformatische Methoden für ganz konkrete Projekte aus vielen Bereichen der Biologie. Der letzte Punkt ist derjenige, der bei uns den größten Teil der Arbeit ausmacht. Obwohl wir selbst am Computer arbeiten, finden bei anderen Projektpartnern die Experimente statt. Diese werden gemeinsam – also auch von uns – geplant. Wir analysieren die gemessenen Daten und bringen sie in eine Form, in der wir ihre Bedeutung wiederum mit den Projektpartnern diskutieren können. Die Vielfalt unserer Projekte reicht dabei von Erregern und Ursachen menschlicher Erkrankungen über mikrobielle Ökosysteme bis hin zu Ameisen, Amöben und sogar der Mumie von "Ötzi".
Es ist Ihnen sogar gelungen, Proteinnetzwerke ausgestorbener evolutionärer Vorläufer zu rekonstruieren. Wie? Und welche Vorläufer sind das?
Rattei: Die Vorfahren, für die wir Proteinnetzwerke rekonstruieren können, ergeben sich direkt aus der Auswahl der für die Berechnung verwendeten heutigen Proteome und Proteinnetzwerke. Nehmen wir z.B. Mensch und Maus, dann rekonstruieren wir das Proteinnetzwerk des letzten gemeinsamen Vorfahren dieser Arten – in diesem Fall also des Vorfahrens aller Säugetiere. In einem zweiten Beispiel, durch Analyse von Mensch, Maus und Hefe, berechnen wir das Proteinnetzwerk des Vorfahrens aller Eukaryoten, d.h. der Organismen mit Zellkern und Organellen. Die Auswahl der Organismen kann leider noch nicht systematisch erfolgen: nur von wenigen Modellorganismen gibt es überhaupt ausreichend Daten zu den heutigen Proteinnetzwerken. Daher war bereits die Analyse der sieben Arten in unserer Arbeit ein großer Fortschritt gegenüber bisherigen Studien.
Gelten die von Ihnen entdeckten Mechanismen auch für den Menschen?
Rattei: Ja, unbedingt. Das Genom des Menschen ist heute das am besten untersuchte. Was aber nicht heißt, dass wir es am besten verstehen. Es ist sehr komplex – beispielsweise existieren von den meisten Proteinen verschiedene Varianten, die aus dem gleichen Gen in verschiedenen Geweben nach einem unterschiedlichen Bauplan gebildet werden. Wir würden gern verstehen, was dies für die Interaktion mit anderen Proteinen bedeutet und welche Mutationen eine Auswirkung darauf haben. Unsere Ergebnisse sind wichtig, um durch den Vergleich der menschlichen Proteine mit denen anderer Arten derartige Fragen zu beantworten.
Ihre Forschungen scheinen für verschiedenste Fachrichtungen von Bedeutung zu sein. Täuscht dieser Eindruck?
Rattei: Nein, keinesfalls. Die Bioinformatik ist eine methodenorientierte Querschnittsdisziplin, mit Bedeutung für praktisch alle Bereiche der Biologie. Es gibt ja kaum einen Zweig der Biologie, in dem molekulare Daten keine Bedeutung hätten. Unsere Forschungen sind ein wichtiger Teil der sogenannten "Systembiologie", die in der Datenanalyse und Modellierung unverzichtbar sind. Die zahlreichen Kooperationen unseres Teams mit Forschern im In- und Ausland dokumentieren dies eindrucksvoll.
Blicken wir einige Jahre in die Zukunft. Was hoffen Sie dann, mehr zu wissen?
Rattei: Die wichtigste Aufgabe der Bioinformatik ist die Bewältigung der Komplexität in der Biologie. Es ist vergleichsweise einfach geworden, molekulare Daten zu messen. Um diese jedoch zu interpretieren, muss das komplexe Zusammenspiel der Moleküle in den Zellen durch Theorien verstanden werden. Da gibt es noch viel Arbeit, denn momentan verwenden wir molekulare Daten ja vor allem, um den molekularen Aufbau der Organismen überhaupt zu beschreiben und den Teilen Funktionen zuzuordnen.
In einigen Jahren wollen wir an dem Punkt sein, an dem die Meteorologie heute schon ist: Durch systematische Messung und leistungsfähige Theorien können gute Vorhersagen über die nahe Zukunft gemacht werden. Wenn wir künftig also das Erbgut von Krankheitserregern oder die molekulare Zusammensetzung menschlicher Zellen überwachen, dann könnten wir Ausbrüche von Infektionen (wie z.B. der EHEC-Epidemie 2011) oder das Entstehen von Krebs im Frühstadium irgendwann mit guter Genauigkeit vorhersagen. (red)



