Seeanemonen zeigen Ähnlichkeiten zu Tieren und Pflanzen
| 18. März 2014Evolutionsbiologe Ulrich Technau und sein Team von der Universität Wien fanden heraus, dass die Seeanemone einerseits eine ähnlich komplexe "Gen-Landkarte" besitzt wie tierische Modellorganismen, zugleich aber auch die Art der Proteinregulation mit Pflanzen gemeinsam hat.
Wie wir aussehen, welche Form wir haben und wie unser Körper funktioniert, ist neben Umwelteinflüssen weitgehend das Werk unserer Gene. Diese sind jedoch selten Einzelspieler, vielmehr wirken sie im Team und regulieren sich in ihrer Aktivität und Expression gegenseitig in gen-regulatorischen Netzwerken.
Einfache Organismen mit komplexem Genmaterial
Im letzten Jahrzehnt haben Genomsequenzierungen von Menschen und vielen Tieren gezeigt, dass anatomisch einfache Organismen wie die Seeanemone erstaunlicherweise ein ähnlich komplexes Genrepertoire wie höhere Modellorganismen aufweisen. Dies legte den Schluss nahe, dass es nicht die An- oder Abwesenheit einzelner Gene ist, die über Unterschiede der morphologischen Komplexität entscheidet. WissenschafterInnen entwickelten die Hypothese, dass genregulatorische Netzwerke von simplen Organismen wie der Seeanemone einfacher gestrickt sind als die des Menschen oder anderer komplexer Tiere.
Sprache der Genetik
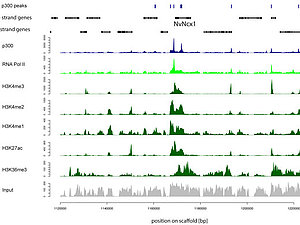
Ein Maß für die Komplexität von Genregulation ist die Verteilung und Dichte von regulatorischen Sequenzen im Genom. An diesen Motiven, "Enhancer" und "Promotoren" genannt, können sich sogenannte Transkriptionsfaktoren mit der DNA in spezifischer Weise binden und die Expression von Zielgenen regulieren. "Diese kurzen Motive in einem Ozean von Nukleotiden zu finden, ist jedoch alles andere als trivial", erklärt Ulrich Technau, Professor am Department für Molekulare Evolution und Entwicklung der Universität Wien.
Während die Gene also gewissermaßen die Worte in der Sprache der Genetik sind, stellen die "Enhancer" und "Promotoren" die Grammatik dar. Diese genregulatorischen Sequenzen korrelieren mit bestimmten chemischen, epigenetischen Modifikationen der mit der DNA überall verwobenen Histon-Proteine, dem Chromatin. Durch eine ausgefeilte molekularbiologische Methode, der Chromatin-Immunpräzipitation, konnte die Hertha-Firnberg-Stipendiatin Michaela Schwaiger in der Gruppe von Ulrich Technau die "Enhancer" und "Promotoren" im gesamten Genom der Seeanemone identifizieren und mit den Karten komplexer Organismen vergleichen.
| Identifikation der verschiedenen Chromatinmodifikationen (Foto: M. Schwaiger). |
|---|
Genregulation vergleichbar mit tierischen Organismen
"Da die Seeanemone eine ähnlich komplexe 'Landkarte' von genregulatorischen Elementen besitzt wie die Fruchtfliege oder andere tierische Modellorganismen, liegt für uns der Schluss nahe, dass dieses Prinzip von komplexer Genregulation auf gemeinsame Vorfahren von Mensch, Fliege und Seeanemone zurückgeht", so Michaela Schwaiger.
MicroRNAs wichtig für Entwicklungsprozesse beim Menschen …
Neben der Kontrolle der Transkription von DNA in RNA kann die Genexpression aber auch noch danach, auf der post-transkriptionellen Ebene, d.h. bei der Übersetzung der RNA in Proteine, reguliert werden. Hierbei spielen "microRNAs" eine wichtige Rolle. MicroRNAs sind kurze RNAs, die an Ziel-RNAs binden und ihre Translation hemmen können. In den letzten Jahren hat man bei vielen Tieren 100-200, beim Menschen sogar über 1000 verschiedene miRNAs identifiziert, von denen etliche eine wichtige Rolle im Stoffwechsel und vor allem in Entwicklungsprozessen spielen.
Mutationen in manchen miRNAs sind mit schweren Fehlentwicklungen und auch Krebs assoziiert. Jede miRNA kann in sequenz-spezifischer Weise an bis zu 100 verschiedene RNAs binden. "Man geht daher davon aus, dass 30-50 Prozent aller menschlichen protein-kodierenden Transkripte durch miRNAs reguliert werden", erklärt Ulrich Technau. Unklar ist jedoch, wie tierische miRNAs in der Evolution entstanden sind.
… und bei Pflanzen
miRNAs hat man auch in Pflanzen entdeckt, doch man geht davon aus, dass sie unabhängig entstanden sind, weil sie (1) keinerlei Sequenzähnlichkeit zu tierischen miRNAs haben, (2) einen anderen Entstehungsweg haben und (3) einen gänzlich anderen Wirkmechanismus haben als die bislang untersuchten Tiere: Pflanzliche miRNAs binden mit hoher Sequenzspezifität an sehr wenige Ziel-RNAs und leiten mithilfe von Argonaute-Proteinen die spezifische Spaltung der RNA ein. In Kollaboration mit amerikanischen, französischen und norwegischen Gruppen haben Ulrich Technau und sein Team insgesamt 87 miRNAs aus der Seeanemone isolieren können.
"Die Seeanemonen-microRNAs haben alle Charakteristika von pflanzlichen microRNAs: Sie weisen eine fast perfekte Komplementarität zu ihren Ziel-RNAs auf, die daraufhin dann gespalten und nicht wie in anderen Tieren gehemmt werden", erklärt Erstautor und Marie-Curie Stipendiat Yehu Moran. Zudem fand Moran zusammen mit David Fredman und Daniela Praher das Protein HYL-1, das bei Pflanzen essenziell für die microRNA Biogenese ist und das man zuvor für pflanzenspezifisch hielt.
Charakteristika von Pflanzen
Die große Überraschung war aber, dass die Seeanemonen-miRNA alle Charakteristika von pflanzlichen miRNAs haben: Sie weisen eine fast perfekte Komplementarität zu ihren Ziel-RNAs auf, die daraufhin dann gespalten und nicht wie in anderen Tieren gehemmt werden. Zudem fanden die Forscher ein Protein, HYL-1, dass bei Pflanzen essenziell für die Biogenese der miRNAs ist und dass man zuvor in keinem der tierischen Modellorganismen gefunden hat. Vergleicht man die Sequenzen der miRNAs, so findet man je eine miRNA mit Ähnlichkeit zu einer pflanzlichen und tierischen miRNA.
Nachdem also das Genom, das Genrepertoire und die Genregulation auf DNA-Ebene der Seeanemone dem der Wirbeltiere erstaunlich ähnlich ist, ist die post-transkriptionelle Regulation so wie in Pflanzen und geht vermutlich auf einen gemeinsamen Vorfahren mit Pflanzen zurück. Diese Befunde sind der erste qualitative Unterschied zwischen den Nesseltieren und den "höheren" Tieren und geben einen Einblick, wie wichtige Ebenen der Genregulation unterschiedlich evolvieren können. (af)
Das Paper "Evolutionary conservation of the eumetazoan gene regulatory landscape" (AutorInnen: M. Schwaiger, A. Schönauer, A.F. Rendeiro, C. Pribitzer, A. Schauer, A.F. Gilles, J.B. Schinko, E. Renfer, D. Fredman, und U. Technau) erschien am 18. März 2014 in Genome Research.
Das Paper "Cnidarian microRNAs frequently regulate their targets by cleavage. In: Genome Research" (AutorInnen: Y. Moran, D. Fredman, D. Praher, L.M. Xi, Z. Meng Wee, F. Rentzsch, P. Zamore, U. Technau, und H. Seitz) erschien am 18. März 2014 in Genome Research.